| Команда | Описание |
| fastqc chr14.1.fastq | Анализ качества чтений. |
| java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr14.1.fastq chr14.1_trimmed.fastq TRAILING:20 MINLEN:50 | Очистка чтений. Обрезали с конца нуклеотды с качеством ниже 20, и убрали все последовательности длиной менее 50 нк |
| hisat2 -x chr14_index -U chr14.1_trimmed.fastq -S chr14.1_align.sam --no-softclip --summary-file hisa_chr14.1_final.txt | Триммированные чтения картированы на геном. -х – путь к индексу -U– путь к чтениям --no-softclip – запрет подрезания чтений --no-spliced-alignment – картирование без разрывов, убираем его, тк в последовательности РНК-транскриптов, откуда вырезаются интроны при созревании, могут картироваться с разрывами. Также результат был сохрнен в отдельный файл. |
| samtools view -b chr14.1_align.sam -o chr14.1_align.bam | Перевод из .sam формата в .bam |
| samtools sort chr14.1_align.bam chr14.1_sorted | Сортировка выравниваний по координате в референсе |
| samtools index chr14.1_sorted.bam | Индексирование |
| htseq-count -f bam -s no -i gene_id -m union chr14.1_sorted.bam gencode.v19.chr_patch_hapl_scaff.annotation.gtf > htseqc_count | Команда для подсчета чтений. Опция -f - формат файла с выравниванием (bam, sam). Опция -s указывает на цепь, по которой были выравнены риды. -i - GFF атрибут, используемый как feature-ID. -m определяет режим работы команды для неоднозначных выравниваний. |
| grep -w 0 -v htseqc_count > results.txt | В текстовый файл записавыются строки, не оканчивающиеся нулем. |
Число чтений до триммирования:18189
Число чтений после триммирования:18134
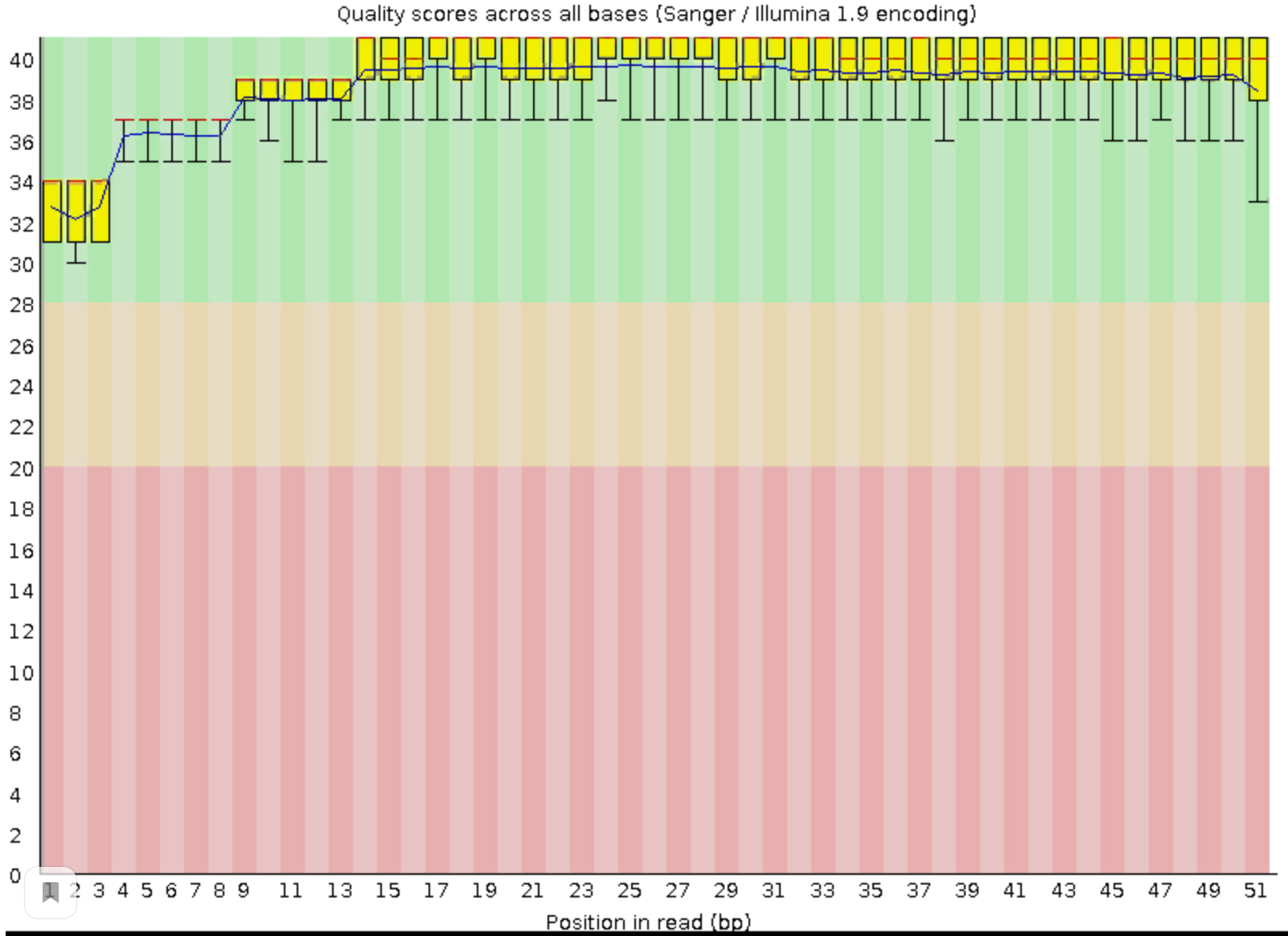
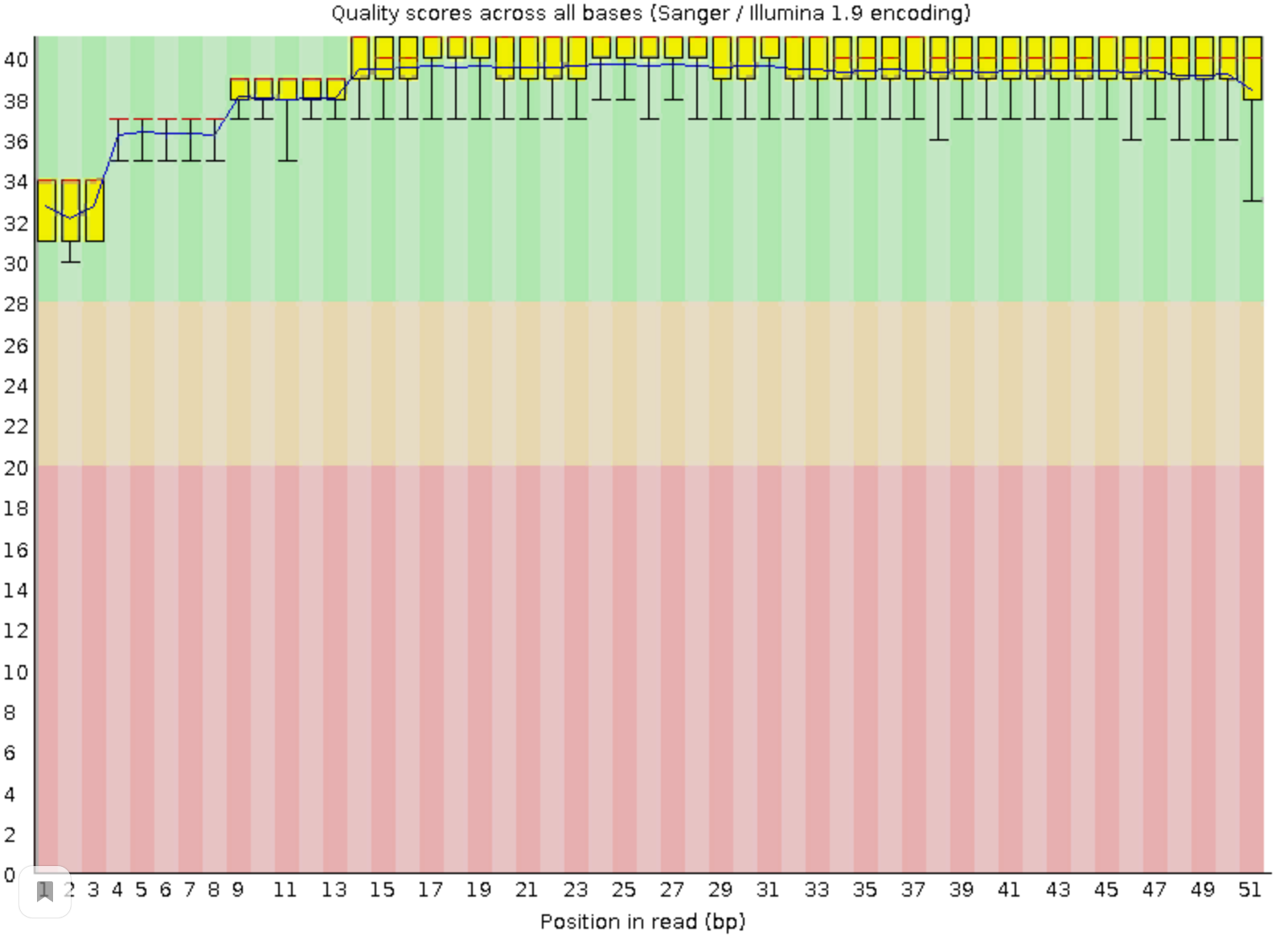
Изначальное качество чтений достаточно хорошее(больше 20). Сравнив качество чтений до и после очистки можно заметить, что ничего особо не изменилось, только в некоторых местах качество стало немного лушче. Поэтому можно сказать, что можно было бы обойтись без триммирования.

По выводу Hisat2 можем сказать, что 99.74% чтений выровнялось ровно 1 раз, 47 чтений не выровнялось ни разу. Качество чтений можно считать высоким.
18134 reads; of these:
18134 (100.00%) were unpaired; of these:
47 (0.26%) aligned 0 times
18087 (99.74%) aligned exactly 1 time
0 (0.00%) aligned >1 times
99.74% overall alignment rate Все чтения легли на один ген. Вывод работы htseq-count:
ENSG00000080824.14 18060 __no_feature 27 __not_aligned 47
Есть 27 прочтений, для которых не определены границы генов, 47 прочтений, которые не картировались.
Ген ENSG00000080824.14 кодирует белок теплового шока hsp90, который защищает клетки, когда они подвергаются воздействию повышенных температур.